Журнал «Здоровье ребенка» Том 17, №4, 2022
Вернуться к номеру
Механізми дії цитоплазматичних мікроРНК. Частина 3. TNRC6-асоційований механізм мікроРНК-опосередкованої деградації мРНК
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
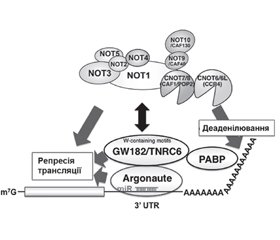
У науковому огляді наведено механізми дії цитоплазматичних мікроРНК, а саме посттранскрипційний сайленсинг: TNRC6-асоційований механізм мікроРНК-опосередкованої деградації мРНК. Для написання статті здійснювався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Відомо, що в цитоплазмі клітини у випадках короткого регіону комплементарності мікроРНК викликають посттранскрипційний сайленсинг, використовуючи перший з основних молекулярних механізмів: TNRC6-асоційований механізм мікроРНК-опосередкованої деградації мРНК. Наведено, що протеїни AGO ссавців містять консервативний мотив m7G-cap-binding protein (відомий як домен MID), необхідний для індукції репресії трансляції, опосередкованої мікроРНК. Після зв’язування даного мотиву протеїну AGO з мікроРНК відбувається рекрутинг протеїнів TNRC6 (GW182), які, у свою чергу, рекрутують різні білки (PABPC1, PAN3 і NOT1), що беруть участь в індукції сайленсингу цільового гена. Автори наводять, що триптофанові залишки (Trp), які розміщені в гідрофобних кишенях протеїнових партнерів TNRC6, зумовлюють високий ступінь афінності й специфічності взаємодій. Науковці вважають, що протеїн TNRC6 при взаємодії з протеїнами AGO може одночасно використовувати три GW/WG повтори (мотив-1, мотив-2 і мотив гака), що розташовані в Argonaute-зв’язуючому домені. Тому протеїн TNRC6 може зв’язуватися одночасно з трьома молекулами AGO. Відомо, що протеїни TNRC6 являють собою PABP-взаємодіючі білки, взаємодія яких з PABP опосередковується консервативним PABP-зв’язуючим мотивом 2. Показано, що протеїни TNRC6 взаємодіють із цитоплазматичним протеїном PABPC1 під час трансляції і стабілізації мРНК. Наведено, що протеїновий комплекс CCR4-NOT являє собою висококонсервативне багатофункціональне мультипротеїнове утворення, що має 3’-5’-екзорибонуклеазну активність, за рахунок якої він і контролює обмін мРНК. Отже, TNRC6-асоційований механізм мікроРНК-опосередкованої деградації мРНК у цитоплазмі клітини викликає посттранскрипційний сайленсинг. При цьому відбувається взаємодія TNRC6 з протеїном PABPC1, рекрутинг протеїнами TNRC6 деаденілюючих комплексів PAN2-PAN3 і CCR4-NOT.
The scientific review presents the mechanisms of action of cytoplasmic miRNAs, namely posttranscriptional silencing: the TNRC6-associated mechanism of miRNA-mediated mRNA degradation. To write the article, information was searched using databases Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. It is known that in the cytoplasm of cells in cases of short region, miRNA complementarities cause posttranscriptional silencing, using the first of the main molecular mechanisms: the TNRC6-associated mechanism of miRNA-mediated mRNA degradation. Mammalian AGO proteins have been shown to contain the conserved m7G-cap-binding protein motif (known as the MID domain), which is required to induce microRNA-mediated translation repression. After binding of this AGO motif to microRNAs, TNRC6 proteins (GW182) are recruited that, in turn, recruits various proteins (PABPC1, PAN3 and NOT1) involved in the induction of the target gene silencing. The authors state that tryptophan residues, which are placed in the hydrophobic pockets of TNRC6 protein partners, cause a high degree of affinity and specificity of interactions. Scientists believe that the TNRC6 protein when interacting with AGO proteins can simultaneously use three GW/WG repeats (motif 1, motif 2 and hook motif), which are located in the Argonaute-binding domain. Therefore, the TNRC6 protein can bind to three AGO molecules simultaneously. TNRC6 proteins are known to be PABP-interacting proteins whose interaction with PABP is mediated by conservative PABP-binding motif 2. TNRC6 proteins have been shown to interact with the cytoplasmic PABPC1 protein during mRNA translation and stabilization. It is shown that the CCR4-NOT protein complex is a highly conserved multifunctional multiprotein formation having 3’-5’-exoribonuclease activity, due to which it controls mRNA metabolism. Thus, the TNRC6-associated mechanism of miRNA-mediated mRNA degradation in the cytoplasm of the cell causes posttranscriptional silencing. In this mechanism, there is an interaction of TNRC6 with PABPC1 protein, recruitment of deadenylating complexes PAN2-PAN3 and CCR4-NOT by the TNRC6 proteins.
мікроРНК; протеїни TNRC6; протеїни PABPC1; деаденілюючий комплекс PAN2-PAN3; деаденілюючий комплекс CCR4-NOT; огляд
microRNA; miRNA; miR; TNRC6 proteins; PABPC1 proteins; deadenylating complex PAN2-PAN3; deadenylating complex CCR4-NOT; review
Вступ
/45.jpg)
Висновки
- Azzouz N., Panasenko O.O., Deluen C. et al. Specific roles for the Ccr4-Not complex subunits in expression of the genome. RNA. 2009 Mar. 15(3). 377-83. doi: 10.1261/rna.1348209.
- Bawankar P., Loh B., Wohlbold L. et al. NOT10 and C2orf29/NOT11 form a conserved module of the CCR4-NOT complex that docks onto the NOT1 N-terminal domain. RNA Biol. 2013 Feb. 10(2). 228-44. doi: 10.4161/rna.23018.
- Boland A., Chen Y., Raisch T. et al. Structure and assembly of the NOT module of the human CCR4-NOT complex. Nat. Struct. Mol. Biol. 2013 Nov. 20(11). 1289-97. doi: 10.1038/nsmb.2681.
- Braun J.E., Huntzinger E., Izaurralde E. The role of GW182 proteins in miRNA-mediated gene silencing. Adv. Exp. Med. Biol. 2013. 768. 147-63. doi: 10.1007/978-1-4614-5107-5_9.
- Bulbrook D., Brazier H., Mahajan P. et al. Tryptophan-Mediated Interactions between Tristetraprolin and the CNOT9 Subunit Are Required for CCR4-NOT Deadenylase Complex Recruitment. J. Mol. Biol. 2018 Mar 2. 430(5). 722-736. doi: 10.1016/j.jmb.2017.12.018.
- Chekulaeva M., Mathys H., Zipprich J.T. et al. miRNA repression involves GW182-mediated recruitment of CCR4-NOT through conserved W-containing motifs. Nat. Struct. Mol. Biol. 2011 Oct 7. 18(11). 1218-26. doi: 10.1038/nsmb.2166.
- Chen C.Y., Shyu A.B. Mechanisms of deadenylation-dependent decay. Wiley Interdiscip. Rev. RNA. 2011 Mar-Apr. 2(2). 167-83. doi: 10.1002/wrna.40.
- Christie M., Boland A., Huntzinger E. et al. Structure of the PAN3 pseudokinase reveals the basis for interactions with the PAN2 deadenylase and the GW182 proteins. Mol. Cell. 2013 Aug 8. 51(3). 360-73. doi: 10.1016/j.molcel.2013.07.011.
- Collart M.A. The Ccr4-Not complex is a key regulator of eukaryotic gene expression. Wiley Interdiscip. Rev. RNA. 2016 Jul. 7(4). 438-54. doi: 10.1002/wrna.1332.
- Collart M.A. The Not4 RING E3 Ligase: A Relevant Player in Cotranslational Quality Contro. ISRN Mol. Biol. 2013 Jan 21. 2013:548359. doi: 10.1155/2013/548359.
- Collart M.A., Panasenko O.O. The Ccr4-Not Complex: Architecture and Structural Insights. Subcell. Biochem. 2017. 83. 349-379. doi: 10.1007/978-3-319-46503-6_13.
- Corley M., Burns M.C., Yeo G.W. How RNA-Binding Proteins Interact with RNA: Molecules and Mechanisms. Mol Cell. 2020 Apr 2. 78(1). 9-29. doi: 10.1016/j.molcel.2020.03.011.
- Das S. Unraveling the CNOT: A new player in the autophagy-cell death nexus. Sci Signal. 2018 Feb 6. 11(516). pii: eaar6364. doi: 10.1126/scisignal.aar6364.
- De Keuckelaere E., Hulpiau P., Saeys Y. et al. Nanos genes and their role in development and beyond. Cell. Mol. Life Sci. 2018 Jun. 75(11). 1929-1946. doi: 10.1007/s00018-018-2766-3.
- Dimitrova L.N., Kuroha K., Tatematsu T., Inada T. Nascent peptide-dependent translation arrest leads to Not4p-mediated protein degradation by the proteasome. J. Biol. Chem. 2009 Apr 17. 284(16). 10343-52. doi: 10.1074/jbc.M808840200.
- Elkayam E., Faehnle C.R., Morales M. et al. Multivalent Recruitment of Human Argonaute by GW182. Mol. Cell. 2017 Aug 17. 67(4). 646-658.e3. doi: 10.1016/j.molcel.2017.07.007.
- Fabian M.R., Sonenberg N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC. Nat. Struct. Mol. Biol. 2012 Jun 5. 19(6). 586-93. doi: 10.1038/nsmb.2296.
- Geissler R., Grimson A. A position-specific 3’UTR sequence that accelerates mRNA decay. RNA Biol. 2016 Nov. 13(11). 1075-1077. Doi: 10.1080/15476286.2016.1225645.
- Ghidini A., Cléry A., Halloy F., Allain FHT, Hall J. RNA-PROTACs: Degraders of RNA-Binding Proteins. Angew. Chem. Int. Ed. Engl. 2021 Feb 8. 60(6). 3163-3169. doi: 10.1002/anie.202012330.
- Han K., Chen S., Cai M. et al. Nanos3 not nanos1 and nanos2 is a germ cell marker gene in large yellow croaker during embryogenesis. Comp. Biochem. Physiol. B. Biochem. Mol. Biol. 2018 Apr. 218. 13-22. doi: 10.1016/j.cbpb.2018.01.002.
- Inada T., Makino S. Novel roles of the multi-functional CCR4-NOT complex in post-transcriptional regulation. Front. Genet. 2014 May 20. 5. 135. doi: 10.3389/fgene.2014.00135.
- Jadhav G.P., Kaur І., Maryati M. et al. Discovery, synthesis and biochemical profiling of purine-2,6-dione derivatives as inhibitors of the human poly(A)-selective ribonuclease Caf1. Bioorg. Med. Chem. Lett. 2015 Oct 1. 25(19). 4219-24. doi: 10.1016/j.bmcl.2015.07.095.
- James N., Landrieux E., Collart M.A. A SAGA-independent function of SPT3 mediates transcriptional deregulation in a mutant of the Ccr4-not complex in Saccharomyces cerevisiae. Genetics. 2007 Sep. 177(1). 123-35. Doi: 10.1534/genetics.107.076299.
- Kuzuoglu-Öztürk D., Huntzinger E., Schmidt S., Izaurralde E. The Caenorhabditis elegans GW182 protein AIN-1 interacts with PAB-1 and subunits of the PAN2-PAN3 and CCR4-NOT deadenylase complexes. Nucleic Acids Res. 2012 Jul. 40(12). 5651-65. doi: 10.1093/nar/gks218.
- Lemay J.F., Lemieux C., St-André O., Bachand F. Crossing the borders: poly(A)-binding proteins working on both sides of the fence. RNA Biol. 2010 May-Jun. 7(3). 291-5. PMID: 20400847.
- Meijer H.A., Kong Y.W., Lu W.T. et al Translational repression and eIF4A2 activity are critical for microRNA-mediated gene regulation. Science. 2013 Apr 5. 340(6128). 82-5. doi: 10.1126/science.1231197.
- Mulder K.W., Brenkman A.B., Inagaki A. et al. Regulation of histone H3K4 tri-methylation and PAF complex recruitment by the Ccr4-Not complex. Nucleic Acids Res. 2007. 35(7). 2428-39. Doi: 10.1093/nar/gkm175.
- Panasenko O.O. The role of the E3 ligase Not4 in cotranslational quality control. Front. Genet. 2014 May 19. 5. 141. doi: 10.3389/fgene.2014.00141.
- Pashler A.L., Towler B.P., Jones C.I., Newbury S.F. The roles of the exoribonucleases DIS3L2 and XRN1 in human disease. Biochem. Soc. Trans. 2016 Oct 15. 44(5). 1377-1384.
- Patial S., Blackshear P.J. Tristetraprolin as a Therapeutic Target in Inflammatory Disease. Trends Pharmacol. Sci. 2016 Oct. 37(10). 811-821. doi: 10.1016/j.tips.2016.07.002.
- Piao X., Zhang X., Wu L., Belasco J.G. CCR4-NOT deadenylates mRNA associated with RNA-induced silencing complexes in human cells. Mol. Cell. Biol. 2010 Mar. 30(6). 1486-94. doi: 10.1128/MCB.01481-09.
- Sanduja S., Blanco F.F., Dixon D.A. The roles of TTP and BRF proteins in regulated mRNA decay. Wiley Interdiscip. Rev. RNA. 2011 Jan-Feb. 2(1). 42-57. doi: 10.1002/wrna.28.
- Schirle N.T., MacRae I.J. The crystal structure of human Argonaute2. Science. 2012 May 25. 336(6084). 1037-40. doi: 10.1126/science.1221551.
- Sgromo A., Raisch T., Backhaus C. et al. Drosophila Bag-of-marbles directly interacts with the CAF40 subunit of the CCR4-NOT complex to elicit repression of mRNA targets. RNA. 2018 Mar. 24(3). 381-395. doi: 10.1261/rna.064584.117.
- Sheu-Gruttadauria J., MacRae I.J. Phase Transitions in the Assembly and Function of Human miRISC. Cell. 2018 May 3. 173(4). 946-957.e16. doi: 10.1016/j.cell.2018.02.051.
- Shirai Y.T., Suzuki T., Morita M. et al. Multifunctional roles of the mammalian CCR4-NOT complex in physiological phenomena. Front. Genet. 2014 Aug 21. 5. 286. doi: 10.3389/fgene.2014.00286.
- Tritschler F., Huntzinger E., Izaurralde E. Role of GW182 proteins and PABPC1 in the miRNA pathway: a sense of déjà vu. Nat. Rev. Mol. Cell. Biol. 2010 May. 11(5). 379-84. doi: 10.1038/nrm2885.
- Ukleja M., Cuellar J., Siwaszek A. et al. The architecture of the Schizosaccharomyces pombe CCR4-NOT complex. Nat. Commun. 2016 Jan 25. 7. 10433. doi: 10.1038/ncomms10433.
- Villanyi Z., Ribaud V., Kassem S. et al. The Not5 subunit of the ccr4-not complex connects transcription and translation. PLoS Genet. 2014 Oct 23. 10(10). e1004569. doi: 10.1371/journal.pgen.1004569.
- Villanyi Z., Collart M.A. Building on the Ccr4-Not architecture. Bioessays. 2016 Oct. 38(10). 997-1002. doi: 10.1002/bies.201600051.
- Wakiyama M., Ogami K., Iwaoka R. et al. MicroRNP-mediated translational activation of nonadenylated mRNAs in a mammalian cell-free system. Genes Cells. 2018 Apr 6. doi: 10.1111/gtc.12580.
- Webster M.W., Stowell J.A.W., Tang T.T.L., Passmore L.A. Analysis of mRNA deadenylation by multi-protein complexes. Methods. 2017 Aug 15. 126:95-104. doi: 10.1016/j.ymeth.2017.06.009.
- Wells M.L., Perera L., Blackshear P.J. An Ancient Family of RNA-Binding Proteins: Still Important! Trends Biochem. Sci. 2017 Apr. 42(4). 285-296. doi: 10.1016/j.tibs.2016.12.003.
- Wiederhold K., Passmore L.A. Cytoplasmic deadenylation: regulation of mRNA fate. Biochem. Soc. Trans. 2010 Dec. 38(6). 1531-6. doi: 10.1042/BST0381531.
- Wolf J., Valkov E., Allen M.D. et al. Structural basis for Pan3 binding to Pan2 and its function in mRNA recruitment and deadenylation. EMBO J. 2014 Jul 17. 33(14). 1514-26. doi: 10.15252/embj.201488373.
- Wolf J., Passmore L.A. mRNA deadenylation by Pan2-Pan3. Biochem. Soc. Trans. 2014 Feb. 42(1). 184-7. doi: 10.1042/BST20130211.
- Xu K., Bai Y., Zhang A. et al. Insights into the structure and architecture of the CCR4-NOT complex. Front Genet. 2014 May 16. 5. 137. doi: 10.3389/fgene.2014.00137.
- Zhang H., Sheng C., Yin Y. et al. PABPC1 interacts with AGO2 and is responsible for the microRNA mediated gene silencing in high grade hepatocellular carcinoma. Cancer Lett. 2015 Oct 10. 367(1). 49-57. doi: 10.1016/j.canlet.2015.07.010.

/44.jpg)
/45_2.jpg)
/46.jpg)
/46_2.jpg)
/47.jpg)
/48.jpg)