Журнал «Здоровье ребенка» Том 17, №1, 2022
Вернуться к номеру
Механізми дії внутрішньоядерних мікроРНК. Частина 2. МікроРНК-опосередкована регуляція нкРНК-транскриптому й альтернативного сплайсингу
Авторы: Абатуров О.Є., Бабич В.Л.
Дніпровський державний медичний університет, м. Дніпро, Україна
Рубрики: Педиатрия/Неонатология
Разделы: Справочник специалиста
Версия для печати
У науковому огляді наведені механізми дії внутрішньоядерних мікроРНК, а саме мікроРНК-опосередкована регуляція нкРНК-транскриптому й альтернативного сплайсингу. Для написання статті виконувався пошук інформації з використанням баз даних Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka. Підкреслено, що істотний вплив на склад транскриптому забезпечує мікроРНК у комплексі RISC, що індукують посттранскрипційну деградацію довгих нкРНК, локалізованих в ядрі клітини. Науковці вважають, що довгі нкРНК беруть участь в епігенетичній регуляції сайленсингу генів за допомогою ремоделювання хроматину. Зазначено, що у розвитку деяких захворювань печінки беруть активну участь довгі нкРНК. Надана інформація про те, що найважливішим механізмом розширення спектра транскриптому у клітині є альтернативний сплайсинг пре-мРНК. Авторами наведені результати наукових досліджень, які свідчать про те, що пре-мРНК більше 90 % генів людини піддаються альтернативному сплайсингу. Зазначено, що сплайсинг виконує спеціалізоване макромолекулярне утворення — супрасплайсосома, яка являє собою мегакомплекс (21 MDA) ядерного рибонуклепротеїну. Показано, що науковцями запропоновані дві моделі епігенетичної регуляції сплайсингу: кінетична модель і модель рекрутингу. Автори розкривають основні положення цих моделей. МікроРНК беруть активну участь у сплайсингу. В основі захворювань печінки можуть лежати дефіцит факторів сплайсингу і дерегуляція альтернативного сплайсингу, зумовлені дією мікроРНК. Порушення альтернативного сплайсингу, які стимулюють проліферацію, запобігають апоптозу та підтримують трансформацію клітин, є патогномонічним явищем при злоякісних пухлинних процесах. Таким чином, складовими механізмами дії внутрішньоядерних мікроРНК є змінювання транскриптому нкРНК та участь у регуляції альтернативного сплайсингу. МікроРНК-опосередкована регуляція стабільності довгих нкРНК обумовлює зміну спектра активності експресії епігенетично регульованих генів. Довгі нкРНК беруть активну участь у розвитку деяких захворювань печінки. Альтернативний сплайсинг є невід’ємною частиною диференціювання клітин і сприяє становленню специфічності тканини. Альтернативний сплайсинг і генерація різноманітних ізоформ протеїнів призводять до молекулярних наслідків, які зумовлюють розвиток різних патологічних станів.
The scientific review presents the mechanisms of action of intranuclear miRNAs, namely microRNA-mediated regulation of the non-coding RNA (ncRNA) transcriptome and alternative splicing. To write the article, information was searched using Scopus, Web of Science, MedLine, PubMed, Google Scholar, EMBASE, Global Health, The Cochrane Library, CyberLeninka databases. It is emphasized that a significant effect on the composition of the transcriptome is provided by microRNAs in the RNA-induced silencing complex that induce posttranscriptional degradation of long ncRNAs localized in the cell nucleus. Scientists believe that long ncRNAs are involved in the epigenetic regulation of gene silencing through chromatin remodeling. It is shown that long ncRNAs are actively involved in the development of some liver diseases. It is reported that the most important mechanism for expanding the spectrum of the transcriptome in the cell is the alternative splicing of pre-mRNA. The authors present the results of scientific studies that show that pre-mRNA of more than 90 % of human genes are subjected to alternative splicing. It is presented that splicing is performed by a specialized macromolecular formation — suprasplicesome, which is a megacomplex (21 MDA) of nuclear ribonucleroprotein. It is shown that scientists have proposed two models of epigenetic regulation of splicing: kinetic and recruitment one. The authors reveal the main provisions of these models. MicroRNAs are actively involved in splicing. Liver disease may be based on a deficiency of splicing factors and deregulation of alternative splicing caused by the action of miRNAs. Disorders of alternative splicing, which stimulate proliferation, prevent apoptosis and support cell transformation, are a pathognomonic phenomenon in malignant tumors. Thus, the constituent mechanisms of action of intranuclear miRNAs are alteration of the ncRNAs transcriptome and participation in the regulation of alternative splicing. MicroRNA-mediated regulation of the stability of long ncRNAs causes a change in the spectrum of activity of expression of epigenetically regulated genes. Long ncRNAs are actively involved in the development of some liver diseases. Alternative splicing is an integral part of cell differentiation and contributes to the formation of tissue specificity. Alternative splicing and generation of various isoforms of proteins determine molecular consequences that cause the development of various pathological conditions.
мікроРНК; транскриптом; альтернативний сплайсинг; сплайсосома; огляд
microRNA; miRNA; miR; transcriptome; alternative splicing; spliceosome; review
Вступ
МікроРНК-опосередкована регуляція нкРНК-транскриптому
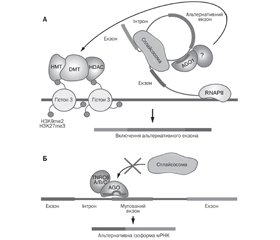
МікроРНК-опосередкована регуляція альтернативного сплайсингу
Висновки
- Дымшиц Г.М., Саблина О.В. «Разорванные» гены и сплайсинг. Вавиловский журнал генетики и селекции. 2014. Т. 18. № 1. С. 71-80.
- Щербик В.В., Бучацкий Л.П. Группа альтернатив и явление сплайсинга. Проблеми екологічної та медичної генетики і клінічної імунології. 2013. № 1 (115). С. 30-38.
- Alló M., Agirre E., Bessonov S. et al. Argonaute-1 binds transcriptional enhancers and controls constitutive and alternative splicing in human cells. Proc. Natl. Acad. Sci U S A. 2014 Nov 4. № 111 (44). Р. 15622-9. doi: 10.1073/pnas.1416858111.
- Alló M., Buggiano V., Fededa J.P. et al. Control of alternative splicing through siRNA-mediated transcriptional gene silencing. Nat. Struct. Mol. Biol. 2009 Jul. № 16(7). Р. 717-24. doi: 10.1038/nsmb.1620.
- Broughton J.P., Pasquinelli A.E. A tale of two sequences: microRNA-target chimeric reads. Genet. Sel. Evol. 2016 Apr 4. № 48. Р. 31. doi: 10.1186/s12711-016-0209-x.
- Carithers L.J., Ardlie K., Barcus M. et al. A Novel Approach to High-Quality Postmortem Tissue Procurement: The GTEx Project. Biopreserv. Biobank. 2015 Oct. № 13 (5). Р. 311-9. doi: 10.1089/bio.2015.0032.
- Chen K., Dai X., Wu J. Alternative splicing: An important mechanism in stem cell biology. World J. Stem. Cells. 2015 Jan 26. № 7 (1). Р. 1-10. doi: 10.4252/wjsc.v7.i1.1.
- Cheng Y., Luo C., Wu W. et al. Liver-Specific Deletion of SRSF2 Caused Acute Liver Failure and Early Death in Mice. Mol. Cell. Biol. 2016 May 16. № 36 (11). Р. 1628-38. doi: 10.1128/MCB.01071-15.
- Fu X.D., Ares M. Jr. Context-dependent control of alternative splicing by RNA-binding proteins. Nat. Rev. Genet. 2014 Oct. № 15 (10). Р. 689-701. doi: 10.1038/nrg3778.
- Grossi I., Salvi A., Abeni E. et al. Biological Function of MicroRNA193a-3p in Health and Disease. Int. J. Genomics. 2017. № 2017. Р. 5913195. doi: 10.1155/2017/5913195.
- Gunewardena S.S., Yoo B., Peng L. et al. Deciphering the Developmental Dynamics of the Mouse Liver Transcriptome. PLoS One. 2015 Oct 23. № 10 (10). Р. e0141220. doi: 10.1371/journal.pone.0141220.
- He Y., Wu Y.T., Huang C. et al. Inhibitory effects of long noncoding RNA MEG3 on hepatic stellate cells activation and liver fibrogenesis. Biochim. Biophys. Acta. 2014 Nov. № 1842 (11). Р. 2204-15. doi: 10.1016/j.bbadis.2014.08.015.
- Iannone C., Valcárcel J. Chromatin’s thread to alternative splicing regulation. Chromosoma. 2013 Dec. № 122 (6). Р. 465-74. doi: 10.1007/s00412-013-0425-x.
- Kalantari R., Chiang C.M., Corey D.R. Regulation of mammalian transcription and splicing by Nuclear RNAi. Nucleic. Acids. Res. 2016 Jan 29. № 44 (2). Р. 524-37. doi: 10.1093/nar/gkv1305.
- Kucherenko M.M., Shcherbata H.R. miRNA targeting and alternative splicing in the stress response — events hosted by membrane-less compartments. J. Cell. Sci. 2018 Feb 14. № 131(4). pii: jcs202002. doi: 10.1242/jcs.202002.
- Li Y., Ren M., Zhao Y. et al. MicroRNA-26a inhibits proliferation and metastasis of human hepatocellular carcinoma by regulating DNMT3B-MEG3 axis. Oncol. Rep. 2017 Jun. № 37 (6). Р. 3527-3535. doi: 10.3892/or.2017.5579.
- Liu Y., Ren F., Luo Y. et al. Down-Regulation of MiR-193a-3p Dictates Deterioration of HCC: A Clinical Real-Time qRT-PCR Study. Med. Sci. Monit. 2015 Aug 11. № 21. Р. 2352-60. doi: 10.12659/MSM.894077.
- Liu Y., Liu X., Lin C. et al. Noncoding RNAs regulate alternative splicing in Cancer. J. Exp. Clin. Cancer Res. 2021. № 40. Р. 11. https://doi.org/10.1186/s13046-020-01798-2
- Ma K., He Y., Zhang H. et al. DNA methylation-regulated miR-193a-3p dictates resistance of hepatocellular carcinoma to 5-fluorouracil via repression of SRSF2 expression. J. Biol. Chem. 2012 Feb 17. № 287 (8). Р. 5639-49. doi: 10.1074/jbc.M111.291229.
- Moehle E.A., Braberg H., Krogan N.J., Guthrie C. Adventures in time and space: splicing efficiency and RNA polymerase II elongation rate. RNA Biol. 2014. № 11(4). Р. 313-9. doi: 10.4161/rna.28646.
- Pan Q., Shai O., Lee L.J., Frey B.J., Blencowe B.J. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing. Nat. Genet. 2008 Dec. № 40 (12). Р. 1413-5. doi: 10.1038/ng.259.
- Peng L., Yoo B., Gunewardena S.S. et al. RNA sequencing reveals dynamic changes of mRNA abundance of cytochromes P450 and their alternative transcripts during mouse liver development. Drug Metab. Dispos. 2012 Jun. № 40 (6). Р. 1198-209. doi: 10.1124/dmd.112.045088.
- Pihlajamäki J., Lerin C., Itkonen P. et al. Expression of the splicing factor gene SFRS10 is reduced in human obesity and contributes to enhanced lipogenesis. Cell. Metab. 2011 Aug 3. № 14 (2). Р. 208-18. doi: 10.1016/j.cmet.2011.06.007.
- Pu M., Chen J., Tao Z., Miao L., Qi X., Wang Y. et al. Regulatory network of miRNA on its target: coordination between transcriptional and post-transcriptional regulation of gene expression. Cell. Mol. Life Sci. 2019. № 76 (3). Р. 441-51. doi: 10.1007/s00018-018-2940-7.
- Saldi T., Cortazar M.A., Sheridan R.M., Bentley D.L. Coupling of RNA Polymerase II Transcription Elongation with Pre-mRNA Splicing. J. Mol. Biol. 2016 Jun 19. № 428 (12). Р. 2623-35. doi: 10.1016/j.jmb.2016.04.017.
- Shefer K., Sperling J., Sperling R. The Supraspliceosome — A Multi-Task Machine for Regulated Pre-mRNA Processing in the Cell Nucleus. Comput. Struct. Biotechnol J. 2014 Sep 28. № 11 (19). Р. 113-22. doi: 10.1016/j.csbj.2014.09.008.
- Sokół E., Kędzierska H., Czubaty A. et al. microRNA-mediated regulation of splicing factors SRSF1, SRSF2 and hnRNP A1 in context of their alternatively spliced 3'UTRs. Exp. Cell. Res. 2018 Feb 15. № 363 (2). Р. 208-217. doi: 10.1016/j.yexcr.2018.01.009.
- Tremblay M.P., Armero V.E., Allaire A. et al. Global profiling of alternative RNA splicing events provides insights into molecular differences between various types of hepatocellular carcinoma. BMC Genomics. 2016 Aug 26. № 17. Р. 683. doi: 10.1186/s12864-016-3029-z.
- Ulitsky I. Interactions between short and long noncoding RNAs. FEBS Lett. 2018 May 10. doi: 10.1002/1873-3468.13085.
- Urbanski L.M., Leclair N., Anczuków O. Alternative-splicing defects in cancer: Splicing regulators and their downstream targets, guiding the way to novel cancer therapeutics. Wiley Interdiscip Rev RNA. 2018 Jul. № 9 (4). Р. e1476. doi: 10.1002/wrna.1476.
- Wang E.T., Sandberg R., Luo S. et al. Alternative isoform regulation in human tissue transcriptomes. Nature. 2008 Nov 27. № 456 (7221). Р. 470-6. doi: 10.1038/nature07509.
- Wang X., Wang J. High-content hydrogen water-induced downregulation of miR-136 alleviates non-alcoholic fatty liver disease by regulating Nrf2 via targeting MEG3. Biol. Chem. 2018 Mar 28. № 399 (4). Р. 397-406. doi: 10.1515/hsz-2017-0303.
- Webster M.W., Stowell J.A.W., Tang T.T.L., Passmore L.A. Analysis of mRNA deadenylation by multi-protein complexes. Methods. 2017 Aug 15. № 126. Р. 95-104. doi: 10.1016/j.ymeth.2017.06.009.
- Yang J.J., Tao H., Deng Z.Y. et al. Non-coding RNA-mediated epigenetic regulation of liver fibrosis. Metabolism. 2015 Nov. № 64 (11). Р. 1386-94. doi: 10.1016/j.metabol.2015.08.004.
- Yeo G., Holste D., Kreiman G., Burge C.B. Variation in alternative splicing across human tissues. Genome Biol. 2004. № 5 (10). R74. doi: 10.1186/gb-2004-5-10-r74.
- Zhao T., Xu J., Liu L. et al. Computational identification of epigenetically regulated lncRNAs and their associated genes based on integrating genomic data. FEBS Lett. 2015 Feb 13. № 589 (4). Р. 521-31. doi: 10.1016/j.febslet.2015.01.013.

/57.jpg)
/58.jpg)